Article
도축장으로부터 분리된 Shigella sonnei 특이적 박테리오파지의 특성 연구
이민정1,
김형우1,
김우주1,
장진욱1,
서정욱1,
곽경민1,
명진종3,
박미경1
,2
,*

Studies on Shigella sonnei-specific bacteriophage isolated from a slaughterhouse
Min-Jeong Lee1,
HyungWoo Kim1,
WooJu Kim1,
JinUk Jang1,
JeongUk Seo1,
KyoungMin Gwak1,
Jinjong Myoung3,
Mi-Kyung Park1
,2
,*

Author Information & Copyright ▼
1School of Food Science and Biotechnology, Kyungpook National University, Daegu 41566Korea
2Food and Bio-industry Research Institute, Kyungpook National University, Daegu 41566Korea
3Korea Zoonosis Research Institute, Chonbuk National University, Jeonju 54896Korea
Copyright ⓒ The Korean Society of Food Preservation. This is an Open-Access article distributed under the terms of the Creative Commons Attribution Non-Commercial License (http://creativecommons.org/licenses/by-nc/3.0/) which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited.
Received: May 05, 2018; Revised: May 29, 2018; Accepted: Jun 04, 2018
Abstract
Continuous outbreaks of Shigella spp. have raised concerns about the lack of rapid and on-site applicable biosensor method for Shigella detection. Since a bacteriophage has recently been employed as an emerging bio-recognition element in biosensor method, Shigella sonnei-specific bacteriophage was isolated and purified from a slaughterhouse with the final concentration of 2.0×1012 PFU/mL in this study. Analysis of purified S. sonnei-specific bacteriophage using transmission electron microscopy indicated that it possessed an icosahedral head with a relatively long non-contractile tail. It was therefore classified as a member of the Siphoviridae family. Head width, head length, and tail length were 69.9±11.2 nm, 77.5±8.8 nm, and 264.4±33.9 nm, respectively. The genomic DNA size of the S. sonnei-specific bacteriophage was determined to be approximately 25 kb by using 0.4% agarose gel electrophoresis. In specificity test with 43 food-associated microorganisms, the S. sonnei-specific bacteriophage exhibited a clear plaque against S. sonnei only. In addition, the S. sonnei-specific bacteriophage was stable within a wide range of pH values (pH 3-11) and temperatures (4-37℃). Thus, the present study demonstrated the excellent specificity and stability of the S. sonnei-specific bacteriophage as a novel bio-recognition element for S. sonnei detection in foods.
Keywords: Shigella sonnei; bacteriophage; bio-recognition; specificity; stability
서 론
Shigella spp.는 그람 음성, 비운동성, 포자를 형성하지 않는 간균으로 주요한 감염병균 중 하나이다. 신선 채소, 우유 및 유제품, 가금류, 어패류, 즉석 편이식품 등이 주요한 원인식품이며(1-3), 섭취 후 12-48시간 내에 설사, 발열, 복부 경련, 탈수를 유발하여 Shigellosis를 야기시킨다(1,4). World Health Organization(WHO)는 전 세계적으로 연간 164.7백만 명이 감염되며, 10-200 정도의 세균수로도 Shigellosis를 유발할 수 있다고 보고하고 있다(1,4). Shigella spp.는 S. sonnei, S. flexneri, S. boydii, S. dysenteriae의 4가지 혈청형으로 나누어지며, 각각의 발병율은 84.9%, 8.7%, 0.3%, 0.1%로 S. sonnei가 압도적인 우위를 차지하고 있다(2-7). 또한 2013년 Centers for Disease Control and Prevention(CDC) 자료에 의하면, 조사한 1,600개의 식품 샘플에서 분리한 Shigella spp.균주의 89%가 항생제에 대해 내성을 갖는다고 보고 하였다(7).
대표적인 검출법의 하나인 배지 기반의 전통적인 검출법은 국제표준기구 Association of Official Analytical Chemists (AOAC), Comité Européen de Normalization(CEN), Association Française de Normalization(AFNOR), Deutsches Institut für Normung(DIN) 등에서 채택된 표준시험방법(gold standard)이나 증균, 선택, 확인 과정을 거치면서 많은 시간과 노력이 소요되는 단점을 가지고 있다. 이러한 단점을 보완하기 위해 Polymerase Chain Reaction(PCR)과 Enzyme Linked Immuno Sorbent Assay(ELISA) 등과 같은 신속검출법이 개발되었고 이들 신속검출법은 비교적 높은 민감성과 특이성을 보여주었다(8-11). 하지만 이들 신속검출법은 DNA 정제와 같은 시료 준비과정이 필요하며, 숙련된 기술과 고가의 장비가 필수적이라는 한계 때문에 식품산업현장에서 손쉽게 사용하기에는 제한적이다(2,8-11). 따라서 1990년대 후반부터 주로 의료용으로 한정적으로 사용되어 왔던 바이오센서 기술이 식중독균 검출에 활용되어 왔다(12). 바이오센서는 대상균에 특이적으로 반응하는 생물학적 수용체(bio-recognition element)와 이들의 결합을 특정한 신호로 변환하여 전달하는 변환기(transducer)로 구성되어 있다(8-10). 따라서, Shigella spp.를 대상으로 한 바이오센서 검출법을 개발하기 위해서는 새로운 생물학적 수용체 탐색에 관한 연구가 선행되어야 한다.
박테리오파지는 숙주 세포를 선별적으로 사멸시키는 세균 바이러스로 전 세계에 1030-1031개 존재하고 있으며(13-14), 높은 특이성뿐만 아니라 온도, pH, 유기용매 등에 대한 높은 안정성, 그리고 특이적인 미생물과의 결합력으로 인해 새로운 생물학적 수용체로 각광받고 있는 실정이다(8,13-16). 따라서, 환경(토양, 온천, 하수 등)이나 식품(신선식품, 돼지고기, 닭고기, 파이, 비스킷, 치즈, 요거트, 홍합, 굴 등)으로부터 다양한 종류의 박테리오파지를 분리하고 있으며, 이를 활용하기 위한 연구가 진행 중이다(12,16). 또한, 미국 FDA는 박테리오파지를 새로운 식품첨가물(Generally Recognized As Safe)로 인정하여 식중독균의 저감화를 위한 “biocontrol agent”로서 이를 식품에 활용하고 있다(16-17). 본 연구에서는 박테리오파지 기반 신속검출법 개발을 위한 선행연구로 도축장 폐수 샘플로부터 S. sonnei에 특이적인 박테리오파지를 분리·정제하여 형태학적 특성을 규명한 후 생물학적 수용체로서의 적용을 검토하기 위해 다양한 균들을 대상으로 하여 그 특이성과 적용을 위한 안정성을 검토하고자 한다.
재료 및 방법
사용균주 및 배양조건
실험에 사용한 균주(Table 1)는 American Type Culture Collection(ATCC), International Vaccine Institute(IVI), Bio-Resource Management for Pathogens National Culture Collection for Pathogens(NCCP), Department of Plant and Food Sciences(DPFS), College of Veterinary Medicine and Research Institute for Veterinary Science, Seoul National University(VMRI)에서 구입 혹은 제공 받아 사용하였다. 각각의 균주 배양을 위해 tryptic soy broth(TSB, Difco Laboratories Inc., Sparks, MD, USA)를 사용하여 37℃에서 110 rpm, 16시간 동안 배양하였다. 원심분리(6,000 rpm, 5분) 후, 균체에 1 mL의 phosphate-buffered saline(PBS, pH 7.4, Life technologies Co., Paisley, UK)를 첨가하였다. 위 단계를 3번 반복하여 순수 균주를 확보하였다. 분광광도계(Optizen 2120UV, Mecasys Co., Ltd., Daejon, Korea)를 이용하여 640 nm에서 균주 현탁액의 흡광도를 측정하고 작성된 표준곡선을 활용하여 각각의 농도를 108 CFU/mL로 하였다.
Table 1.
Specificity of Shigella sonnei-specific bacteriophage against food-associated microorganisms
| Bacterial strain |
Plaque formation1) |
Source2) |
| Shigella sonnei ATCC 9290 |
+ |
ATCC |
| Shigella boydii NCCP 11190 |
- |
NCCP |
| Shigella flexneri 2a strain 2457T |
- |
IVI |
| Aeromonas hydrophila ATCC 7966 |
- |
ATCC |
| Aeromonas hydrophila JUNAH |
- |
VMRI |
| Aeromonas hydrophila SNUFPC-A3 |
- |
VMRI |
| Aeromonas hydrophila SNUFPC-A5 |
- |
VMRI |
| Aeromonas hydrophila SNUFPC-A6 |
- |
VMRI |
| Aeromonas hydrophila SNUFPC-A7 |
- |
VMRI |
| Aeromonas hydrophila SNUFPC-A8 |
- |
VMRI |
| Aeromonas hydrophila SNUFPC-A9 |
- |
VMRI |
| Aeromonas hydrophila SNUFPC-A10 |
- |
VMRI |
| Aeromonas hydrophila SNUFPC-A11 |
- |
VMRI |
| Bacillus cereus ATCC 13061 |
- |
ATCC |
| Bacillus cereus ATCC 14579 |
- |
ATCC |
| Bacillus subtilis ATCC 6633 |
- |
ATCC |
|
Campylobacter jejuni
|
- |
DPFS |
| Escherichia coli ATCC 15144 |
- |
ATCC |
| Escherichia coli ATCC BAA 2192 |
- |
ATCC |
| Escherichia coli ATCC BAA 2196 |
- |
ATCC |
| Escherichia coli O157:H7 ATCC 43895 |
- |
ATCC |
| Klebsiella pneumoniae ATCC 13883 |
- |
ATCC |
| Listeria monocytogenes ATCC 19116 |
- |
ATCC |
| Listeria monocytogenes ATCC 7644 |
- |
ATCC |
| Listeria innucua ATCC 33090 |
- |
ATCC |
| Pseudomonas aeruginosa ATCC 9027 |
- |
ATCC |
|
Salmonella Dublin
|
- |
DPFS |
| Salmonella Enteritidis ATCC 13076 |
- |
ATCC |
| Salmonella Hartford |
- |
DPFS |
| Salmonella Heidelberg |
- |
DPFS |
| Salmonella Mission |
- |
DPFS |
| Salmonella Montevideo |
- |
DPFS |
| Salmonella Newport |
- |
DPFS |
| Salmonella Panama |
- |
DPFS |
| Salmonella Salamae |
- |
DPFS |
| Salmonella Senftenberg |
- |
DPFS |
| Salmonella Typhi |
- |
DPFS |
| Salmonella Typhimurium ATCC 13311 |
- |
ATCC |
| Salmonella Typhimurium ATCC 15812 |
- |
ATCC |
| Staphylococcus aureus ATCC 25923 |
- |
ATCC |
| Vibrio parahaemolyticus ATCC 17802 |
- |
ATCC |
| Vibrio vulnificus |
- |
DPFS |
| Yersinia enterocolitica ATCC 23715 |
- |
ATCC |
Download Excel Table
S. sonnei 특이적 박테리오파지의 분리·정제
영천 ㈜삼세 도축장으로부터 채취한 2개의 도축 폐수 샘플 25 mL를 각각 TSB 225 mL와 1 mL의 S. sonnei ATCC 9290(108 CFU/mL)에 혼합하여 37℃에서 160 rpm, 16시간 동안 배양하였다. 원심분리(8,000 rpm, 10분)한 후 상층액을 0.20 μm cellulose acetate filter(Advantec Tokyo Ltd., Tokyo, Japan)로 여과하였다. 200 μL의 S. sonnei ATCC 9290(108 CFU/mL)와 4 mL의 TA soft agar(4 g agar, 8 g nutrient broth, 5 g NaCl, 0.2 g MgSO4, 0.05 g MnSO4, 0.15 g CaCl2/L)를 혼합하여 TSB 고형 배지(TSA, Difco Laboratories Inc., Sparks, MD, USA)에 부어 굳힌 후, 여과된 박테리오파지 10 μL를 TSB 고형배지 4곳에 분주하였다(dot assay). 희석한 박테리오파지 여과액 100 μL와 200 μL의 S. sonnei ATCC 9290(108 CFU/mL)를 4 mL의 TA soft agar에 혼합하여 TSB 고형배지에 부어 굳힌 후, 37℃에서 16시간 동안 배양하였다(plaque assay). 단일 plaque를 취하여 sodium chloride-magnesium sulphate buffer(SM buffer, 50 mM Tris-HCl, 100 mM NaCl, 10 mM MgSO4, pH 7.5)에 1시간 동안 실온에서 교반하였다. 위 단계를 2번 반복하여 단일 박테리오파지를 분리하였다. 분리된 단일 박테리오파지의 증식을 위해 3 mL의 TA broth에 1% S. sonnei ATCC 9290(108 CFU/mL)를 혼합하여 37℃에서 190 rpm, 2시간 동안 배양하였다. 배양 후, 혼합액을 원심분리(6,000 rpm, 10분)한 후 상층액을 0.20 μm cellulose acetate filter로 여과하였다. 위 단계를 TA broth 양을 증가하면서 반복한 후 최종적으로 여과된 박테리오파지는 10%(w/v) polyethylene glycol과 1 M NaCl 용액을 혼합하여 4℃에서 16시간 동안 정치시킨 후 원심분리(8,000 rpm, 20분) 하였다. SM buffer로 pellet을 현탁한 후, CsCl 밀도 구배 초원심분리(22,000 rpm, 2시간)를 실시하였다. 생성된 박테리오파지 band를 회수하여 냉장 온도에서 6시간 동안 투석하였다. 정제된 박테리오파지의 최종 농도는 plaque assay로 확인하였다.
S. sonnei 특이적 박테리오파지의 형태학적 특성 분석
분리·정제된 S. sonnei 특이적 박테리오파지 10 μL를 carbon-coated copper grid에 올려 4%의 phosphotungstic acid(Sigma-Aldrich Co., St. Louis, MO, USA)로 염색한 후 Transmission electron microscope(TEM, H-7100, Hitachi Ltd., Tokyo, Japan)으로 형태학적 특성을 관찰하였다.
S. sonnei 특이적 박테리오파지의 DNA 분리 및 크기 분석
정제된 S. sonnei 특이적 박테리오파지의 genomic DNA(gDNA)를 NORGEN® bacteriophage DNA isolation kit(Norgen Biotek Co., Thorold, ON, Canada)로 분리·정제하였고, 정제된 gDNA의 농도는 picodrop(Picodrop Ltd., Hinxton, UK)으로 측정하였다. 정제된gDNA의 크기를 확인하기 위해 50 V에서 1.5시간 0.4% 아가로스 겔 전기영동(Takara Bio Ltd., Shiga, Japan)하였다. DNA ladder (GeneRuler High Range DNA ladder, Thermo Fisher Scientific, Inc., Waltham, MA, USA)로 gDNA 크기를 결정하였다.
S. sonnei 특이적 박테리오파지의 특이성 분석
분리·정제된 S. sonnei 특이적 박테리오파지의 특이성 검토를 위해 28종의 식중독균을 각각 TSB에 배양한 후(108 CFU/mL) dot assay법을 이용한 clear zone 생성 여부로 감염숙주 여부를 확인하였다.
S. sonnei 특이적 박테리오파지의 pH 및 온도 안정성 검토
정제된 S. sonnei 특이적 박테리오파지(109 PFU/mL) 100 μL와 다양한 pH 범위를 가진 TSB 900 μL(pH 3, 5, 7, 9, 11)를 혼합한 후 1시간 동안 실온에서 배양하였다. 온도 안전성 검토를 위해 박테리오파지(109 PFU/mL) 100 μL와 SM buffer(pH 7.5) 900 μL를 혼합하여 다양한 온도(4, 22, 37, 45, 50℃)에 1시간 동안 반응시킨 후 배양하였다. 배양 후, plaque assay를 통해 역가를 측정하였다.
결과 및 고찰
S. sonnei 특이적 박테리오파지의 분리·정제
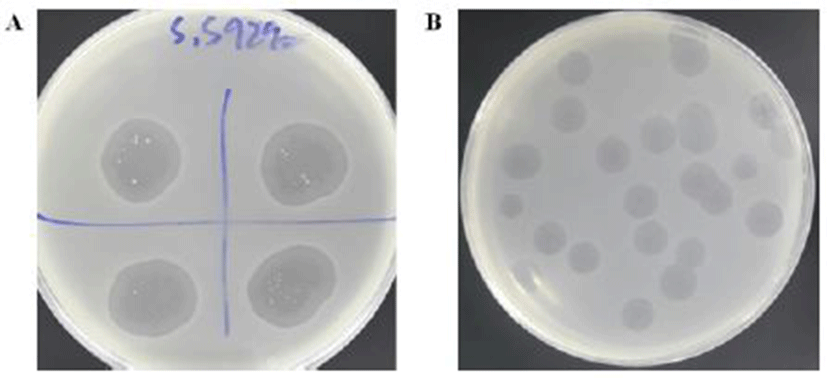
영천 ㈜삼세 도축장으로부터 수집된 도축 폐수 샘플에서 S. sonnei ATCC 9290에 대응하는 2개의 박테리오파지가 분리되었고, 분리된 두 개의 박테리오파지를 대상으로 dot assay를 수행한 결과 clear zone의 크기가 각각 1.88±0.15 cm, 1.83±0.05 cm로 확인되었다. 그 중 clear zone 크기가 크고 선명한(1.88±0.15 cm, Fig. 1A) 박테리오파지를 대상으로 plaque assay를 수행하여 단일의 박테리오파지를 분리하였고(Fig. 1B), 단일의 박테리오파지는 증식과정을 통해 최종적으로 2.0×1012 PFU/mL 농도로 정제되었다. 정제된 단일의 박테리오파지를 S. sonnei 특이적 박테리오파지(S. sonnei-specific bacteriophage)로 명명하였다.
Fig. 1.
Plaques formation of S. sonnei-specific bacteriophage using (A) dot assay and (B) plaque assay.
Download Original Figure
S. sonnei 특이적 박테리오파지의 형태학적 특성 분석
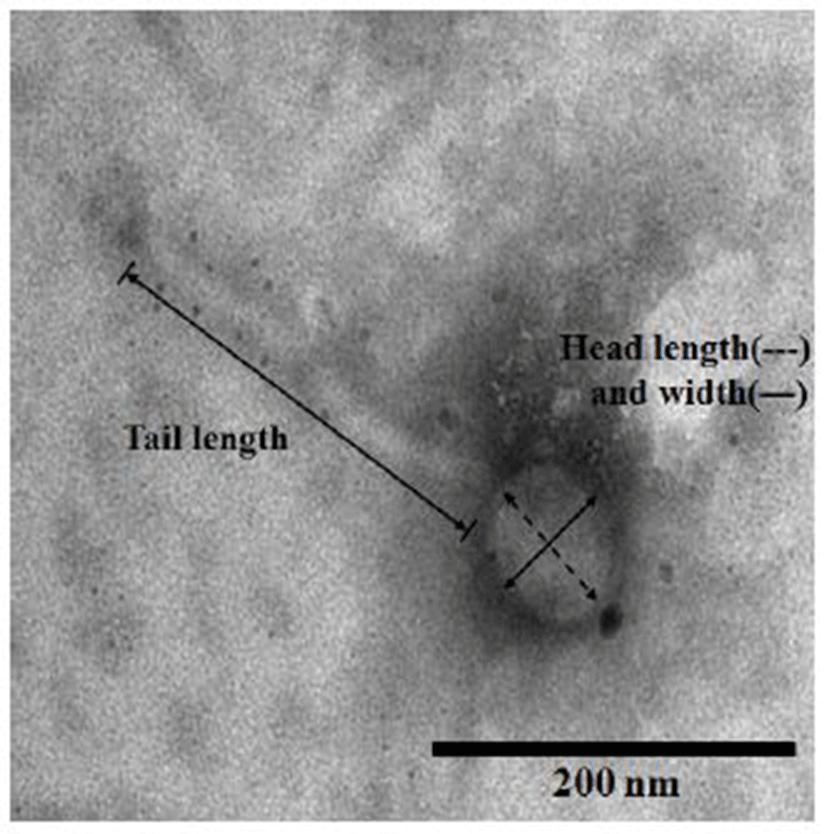
S. sonnei 특이적 박테리오파지의 TEM 분석 결과(Fig. 2), 머리 너비와 길이는 각각 69.9±11.2 nm와 77.5±8.8 nm였으며, 꼬리 길이는 264.4±33.9 nm로 관찰되었다. 이십면체 머리와 비교적 긴 비수축성 꼬리를 가지고 있어, International Committee of Taxonomy of Viruses(ICTV)에서 제시한 분류방법에 따라 Caudoviridae 목의 Siphoviridae에 속함을 확인하였다(18). 기존의 연구결과에 의하면(Table 2), Jun 등(19)은 서울 홍제천 개울물로부터 Myoviridae로 분류되는 S. sonnei와 S. flexneri에 특이적인 한 개의 박테리오파지(pSs-1)를 분리하였고 머리 길이 98±4 nm, 꼬리 길이 120±7 nm로 보고하고 있다. 또한 Faruque 등(20)은 방글라데시 수도 다카의 강 2곳에서 71개의 강물 샘플로부터 Podoviridae로 분류되는 S. dysenteriae 특이적 박테리오파지(SF-9)를 분리하였고, 상당히 짧은 꼬리(TEM 이미지 분석 결과 5 nm로 추정)를 가짐을 확인하였다. 또한 Jun 등(21)은 서울 한강 물 샘플로부터 머리 길이와 꼬리 길이가 각각 73 nm와 103 nm인 Siphoviridae로 분류되는 Shigella spp. 특이적 박테리오파지 즉 pSf-1을 분리하였고, Strauch 등(22)은 설사 환자의 분변으로부터 머리 길이와 꼬리 길이가 약 65 nm와 약 26 nm로 상당히 짧은 꼬리를 가진 S. sonnei CB7888 특이적 박테리오파지를 분리하여 이를 Podoviridae로 분류하였다. 또한 Kim 등(23)은 Myoviridae로 분류되는 S. sonnei 특이적 박테리오파지(SP18)를 분리하였고, 머리 길이 110 nm, 꼬리 길이 50-110 nm로 보고하고 있다. 따라서 본 연구에서 분리된 S. sonnei 특이적 박테리오파지는 기존의 연구된 다수의 연구들과는 달리 Siphoviridae에 속하며 특히 꼬리의 길이가 Siphoviridae로 분류되는 pSf-1보다 거의 두 배 정도 길다는 점에서 기존에 보고된 Shigella spp. 특이적 박테리오파지와는 다른 형태학적 특성을 가지고 있음을 확인 할 수 있었다.
Table 2.
Shigella spp. specific bacteriophages reported in scientific research articles
| Phage name |
Classification |
Morphology |
Stability |
DNA size
(kb) |
References |
| Head length |
Tail length |
pH |
Temp. (℃) |
| S. sonnei-specific bacteriophage |
Siphoviridae
|
77.5 nm |
264.4 nm |
3-11 |
4-37 |
25 |
This study |
| pSs-1 |
Myoviridae
|
98 nm |
120 nm |
- |
- |
160-250 |
(19)
|
| SF-9 |
Podoviridae
|
- |
- |
6-9 |
4-37 |
41 |
(20)
|
| pSf-1 |
Siphoviridae
|
73 nm |
103 nm |
5-9 |
4-50 |
51 |
(21)
|
| 7888 |
Podoviridae
|
65 nm |
26 nm |
- |
- |
63.4 |
(22)
|
| SP18 |
Myoviridae
|
110 nm |
50-110 nm |
- |
- |
170 |
(23)
|
| HY01 |
Myoviridae
|
100 nm |
100 nm |
4-11 |
-20-65 |
167 |
(24)
|
Download Excel Table
S. sonnei 특이적 박테리오파지의 DNA 분리 및 크기 분석
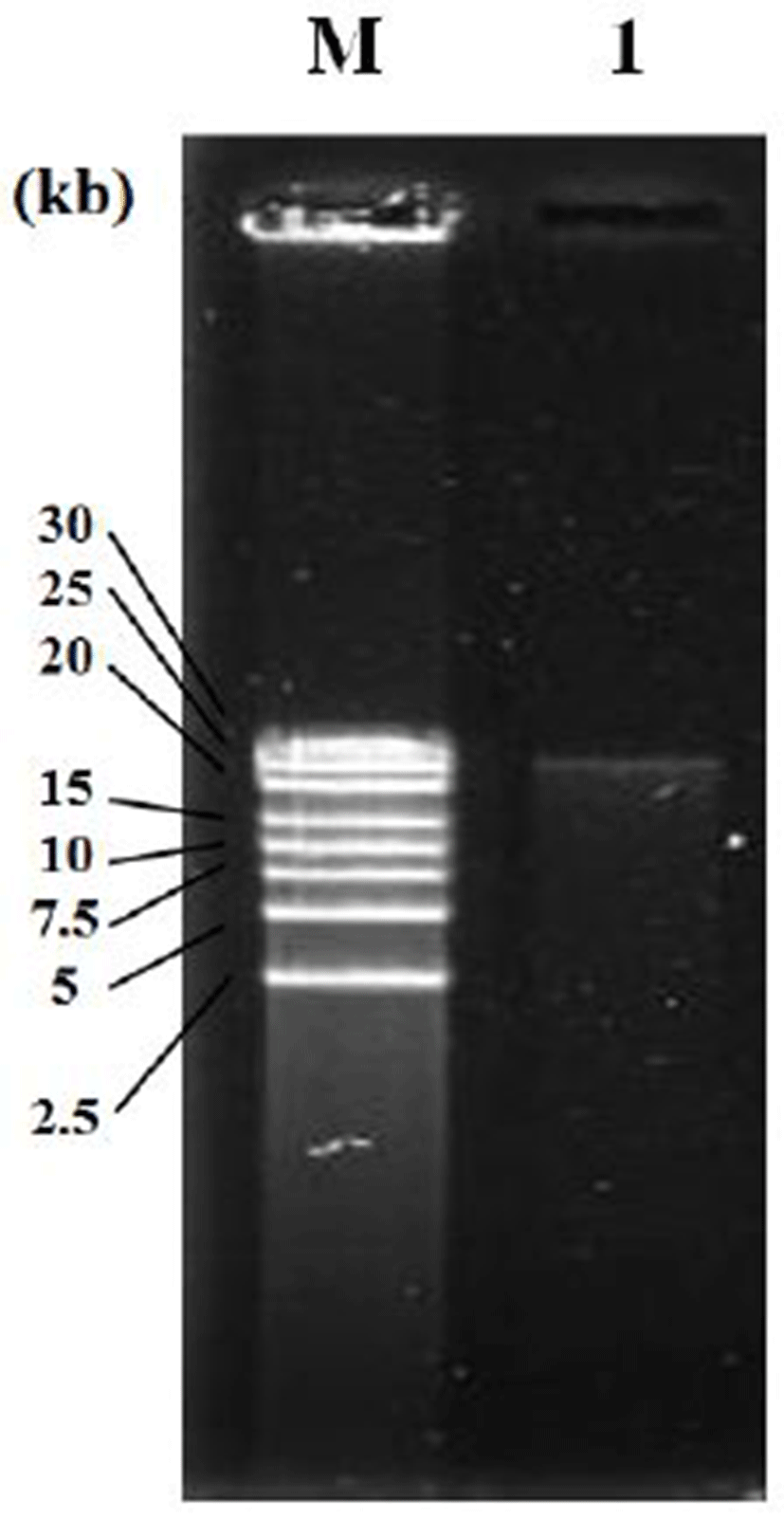
앞의 실험방법으로 분리된 S. sonnei 특이적 박테리오파지를 0.4% 아가로스 겔로 전기영동 한 결과, 2.5 kb ladder를 size marker로 이용하여 약 25 kb DNA 크기의 band를 확인 할 수 있었다(Fig. 3). 기존의 연구결과와 비교해 보면, S. sonnei 특이적 박테리오파지는 약 41 kb 크기인 S. dysenteriae 특이적 박테리오파지(SF-9)(20)와 약 51 kb 크기의 Shigella spp. 특이적 박테리오파지(pSf-1)(21), 그리고 약 63 kb 크기의 S. sonnei CB7888 특이적 박테리오파지와 비교적 비슷한 크기의 DNA를 갖고 있음을 확인 할 수 있었다. 이에 비해 약 160-250 kb 크기의 Shigella spp. 특이적 박테리오파지(pSs-1)(19), 약 167 kb 크기의 S. flexneri와 Escherichia coli O157:H7 특이적 박테리오파지(HY01)(24), 그리고 약 170 kb 크기의 S. sonnei에 특이적 박테리오파지(SP18)(25)보다는 그 크기가 훨씬 작음을 알 수 있었다. 유전자의 크기는 일반적으로 박테리오파지의 분류학상 특성과 상관관계가 있으며, 일반적으로 Siphoviridae과에 속하는 박테리오파지의 DNA 크기가 35-70 kb 임을 감안하다면(13), S. sonnei 특이적 박테리오파지는 상당히 작은 크기의 DNA를 함유하고 있음을 확인 할 수 있었고 이는 추가적인 유전자 염기서열 분석을 통해 박테리오파지의 신규성과 그 크기를 검증할 계획이다.
Fig. 3.
Genomic DNA of S. sonnei-specific bacteriophage using 0.4% agarose gel electrophoresis.
Lane M, 2.5 kb ladder; Lane 1, S. sonnei-specific bacteriophage.
Download Original Figure
S. sonnei 특이적 박테리오파지의 특이성 분석
생물학적 수용체로서 요건을 검토하기 위해 43종의 균을 대상으로 S. sonnei 특이적 박테리오파지의 특이성 검토 결과, S. sonnei 특이적 박테리오파지는 S. sonnei ATCC 9290에만 특이적으로 반응하여 1.88±0.15 cm 크기의 plaque를 형성하였다(Table 1). 하지만, 10종의 Aeromonas hydrophila, 2종의 Bacillus cereus, 1종의 Bacillus subtilis, 1종의 Campylobacter jejuni, 4종의 Escherichia coli, 1종의 Klebsiella pneumoniae, 3종의 Listeria spp., 1종의 Pseudomonas aeruginosa, 13종의 Salmonella spp., 1종의 Staphylococcus aureus, 2종의 Vibrio spp., 1종의 Yersinia enterocolitica에서는 plaque를 형성하지 않았다. 또한, Shigella spp.의 다른 종 즉 S. boydii NCCP 11190, S. flexneri 2a strain 2457T에 대해 특이성을 나타내지 않았다. Jun 등(19)은 특이성 분석을 위해 9개의 Shigella spp.(4종의 S. sonnei, 3종의 S. flexneri, 2종의 S. boydii)를 대상으로 그 중에서 4종의 S. sonnei와 3종의 S. flexneri에 특이적인 박테리오파지를 분리하였다고 기술하였으나 이들 연구는 9종의 Shigella spp.만 제한적으로 사용하였으므로 그 한계를 가지고 있다. 따라서 본 연구에서 분리·정제된 S. sonnei 특이적 박테리오파지는 기존에 보고된 Shigella spp.에 특이적 박테리오파지보다 더욱 우수한 특이성을 갖는 박테리오파지임을 확인할 수 있었다.
S. sonnei 특이적 박테리오파지의 pH 및 온도 안정성
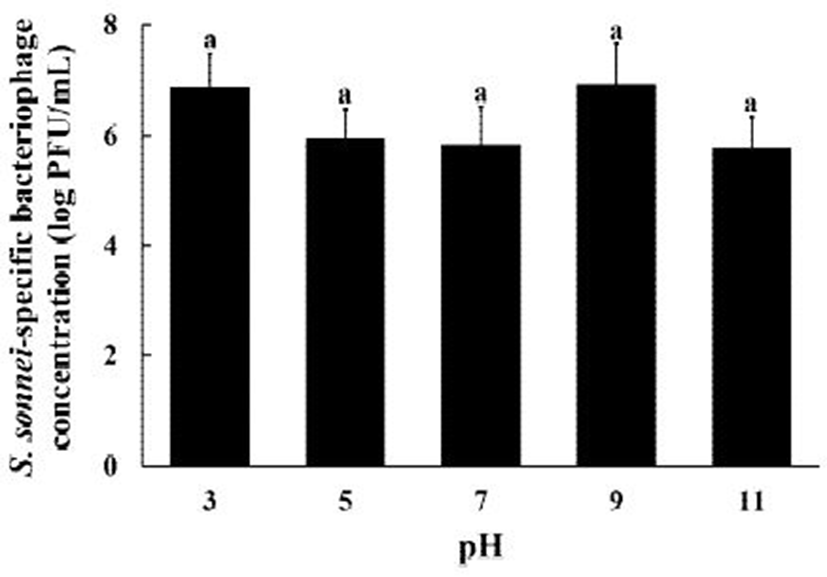
생물학적 수용체로서 센서에 적용되는 박테리오파지는 다양한 환경에 노출되므로 정제된 S. sonnei 특이적 박테리오파지의 pH 및 온도 안정성은 무엇보다 중요한 특성 중의 하나이다(10). 따라서 본 연구에서 S. sonnei 특이적 박테리오파지를 다양한 pH(Fig. 4)와 온도(Fig. 5) 범위에 노출시켜 그 안정성을 검토하였다. Fig. 4에서 보듯이, S. sonnei 특이적 박테리오파지의 농도는 pH 3, 5, 7, 9 그리고 11에서 6.87±0.62 log PFU/mL, 5.94±0.54 log PFU/mL, 5.86±0.66 log PFU/mL, 6.93±0.73 log PFU/mL, 그리고 5.76±0.57 log PFU/mL로 각각 확인되었다. S. sonnei 특이적 박테리오파지의 농도는 pH 3과 pH 9에서 비교적 높게 확인되었으나, 전 범위(pH 3-11)에 걸쳐 유의적인 농도변화는 확인되지 않았다(p>0.05). Faruque 등(20)은 S. dysenteriae 특이적 박테리오파지(SF-9)의 pH 안정성 검토 결과 pH 6-9에서 안정함을 보여주었고, Lee 등(24)은 S. flexneri와 Escherichia coli O157:H7 특이적 박테리오파지(HY01)가 pH 4-11 범위에서 안정함을 보여주었다. 이에 비해 Jun 등(21)은 Shigella spp. 특이적 박테리오파지(pSf-1)가 pH 5-9에서 안정하였으나, pH 3과 pH 11에서는 그 농도가 거의 100% 감소함을 보여주었다. 따라서, S. sonnei 특이적 박테리오파지는 기존의 연구된 다른 Shigella spp. 특이적 박테리오파지보다 넓은 범위에서 우수한 안정성을 보여 주었다.
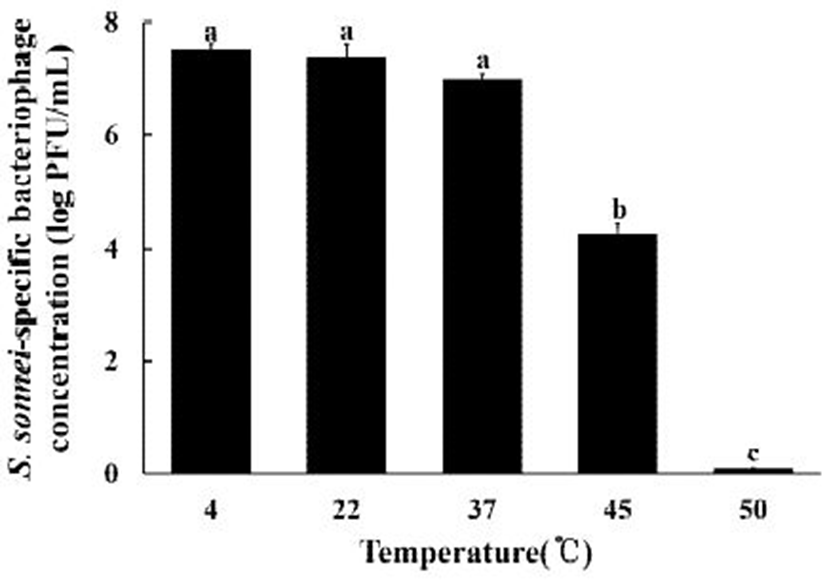
Fig. 5.
Temperature stability of S. sonnei-specific bacteriophage.
The letters (a-c) indicate significantly different means among the group at p<0.05.
Download Original Figure
또한, 온도에 대한 안정성 검토 결과, 4-37℃ 범위에서는 안정적이었으나, 45℃에서는 그 농도가 4.25±0.20 log PFU/mL로 40% 이상 유의적으로 감소하였으며, 50℃에서는 0.10±0.00 log PFU/mL로 거의 98% 이상 유의적으로 감소함을 확인할 수 있었다(p<0.05). 본 연구의 결과는 Shigella spp. 특이적 박테리오파지(pSf-1)가 4℃, 20℃, 그리고 25℃에서는 안정적이었으나 30℃부터 그 농도가 유의적으로 감소하기 시작하여 65℃에서는 거의 100% 감소한다는 Jun 등(21)의 결과보다 높은 온도 안정성을 보여 주었다. 또한, S. dysenteriae 특이적 박테리오파지(SF-9)를 대상으로 한 실험결과 4-37℃에서는 안정함을 보여준 반면, 45℃에서 농도가 유의적으로 감소한다는 유사한 실험결과를 보여 주었다(20). 따라서, S. sonnei 특이적 박테리오파지는 비교적 넓은 범위의 pH와 온도에 안정함을 확인할 수 있었고, 새로운 생물학적 수용체로서 광범위한 pH와 온도를 갖는 다양한 식품에 적용 가능함을 잠재적으로 보여주었다.
요 약
본 연구에서는 영천 ㈜삼세 도축장 폐수 샘플에서 S. sonnei 특이적 박테리오파지를 분리·정제한 후 생물학적 수용체로서의 특성을 검토하였다. Plaque assay를 통해 정제된 박테리오파지의 최종농도는 2.0×1012 PFU/mL로 확인되었고, S. sonnei 특이적 박테리오파지로 명명하였다. TEM 결과 S. sonnei 특이적 박테리오파지는 비수축성의 비교적 긴 꼬리(264.4±33.9 nm)와 이십면체의 머리로 이루어져 있었으며, 머리 너비와 길이는 각각 69.9±11.2 nm와 77.5±8.8 nm로 Siphoviridae과에 속함을 확인하였다. 0.4% 아가로스 겔 전기영동 결과 S. sonnei 특이적 박테리오파지의 DNA 크기는 약 25 kb였으며, 특이성 검토 결과, S. boydii, S. flexneri를 포함한 42개의 균에는 반응하지 않고 S. sonnei에만 특이적으로 반응하여 plaque를 형성하였다. S. sonnei 특이적 박테리오파지의 pH 및 온도 안정성 검토 결과 pH 3-11 그리고 4-37℃에서 모두 높은 안정성을 보여 주었으므로 S. sonnei 특이적 박테리오파지는 신규의 생물학적 수용체로서 S. sonnei를 검출할 수 있는 신속검출법 개발에 활용될 수 있는 충분한 잠재성을 보여주었다.
Acknowledgements
본 연구는 농촌진흥청 과제(PJ012290)의 연구비 지원에 의해 수행되었으며 이에 감사드립니다.
References
Soffer N, Woolston J, Li M, Das C, Sulakvelidze A (2017) Bacteriophage preparation lytic for Shigella significantly reduces Shigella sonnei contamination in various foods. PLoS One, 12, e0175256
SofferN, WoolstonJ, LiM, DasC, SulakvelidzeA. 2017; Bacteriophage preparation lytic for
Shigella significantly reduces
Shigella sonnei contamination in various foods. PLoS One. 12 e0175256


Park MK, Park JW, Wikle HC, Chin BA (2013) Evaluation of phage-based magnetoelastic biosensors for direct detection of Salmonella Typhimurium on spinach leaves. Sens Actuators B, 176, 1134-1140
ParkMK, ParkJW, WikleHC, ChinBA. 2013; Evaluation of phage-based magnetoelastic biosensors for direct detection of
Salmonella Typhimurium on spinach leaves. Sens Actuators B. 176:1134-1140


Petrovski S, Dyson ZA, Seviour RJ, Tillett D (2012) Small but sufficient: The Rhodococcus phage RRH1 has the smallest known Siphoviridae genome at 14.2 kilobases. J Virol, 86, 358-363
PetrovskiS, DysonZA, SeviourRJ, TillettD. 2012; Small but sufficient: The
Rhodococcus phage RRH1 has the smallest known
Siphoviridae genome at 14.2 kilobases. J Virol. 86:358-363


Kotloff KL, Winickoff JP, Ivanoff B, Clemens JD, Swerdlow DL, Sansonetti PJ, Adak GK, Levine MM (1999) Global burden of Shigella infections: implications for vaccine development and implementation of control strategies. Bull W H O, 77, 651-666
KotloffKL, WinickoffJP, IvanoffB, ClemensJD, SwerdlowDL, SansonettiPJ, AdakGK, LevineMM. 1999; Global burden of
Shigella infections: implications for vaccine development and implementation of control strategies. Bull W H O. 77:651-666.

Ahmed AM, Shimamoto T (2015) Molecular characterization of multidrug-resistant Shigella spp. of food origin. Int J Food Microbiol, 194, 78-82
AhmedAM, ShimamotoT. 2015; Molecular characterization of multidrug-resistant
Shigella spp. of food origin. Int J Food Microbiol. 194:78-82


Zafar A, Sabir N, Bhutta ZA (2005) Frequency of isolation of Shigella serogroups/serotypes and their antimicrobial susceptibility pattern in children from slum areas in Karachi. J Pak Med Assoc, 55, 184-188
ZafarA, SabirN, BhuttaZA. 2005; Frequency of isolation of
Shigella serogroups/serotypes and their antimicrobial susceptibility pattern in children from slum areas in Karachi. J Pak Med Assoc. 55:184-188.

Park MK, Oh JH (2012) Rapid detection of E. coli O157:H7 on turnip greens using a modified gold biosensor combined with light microscopic imaging system. Food Sci, 77, M127-M134
ParkMK, OhJH. 2012; Rapid detection of
E. coli O157:H7 on turnip greens using a modified gold biosensor combined with light microscopic imaging system. Food Sci. 77:M127-M134


Hiremath N, Guntupalli R, Vodyanoy V, Chin BA, Park MK (2015) Detection of methicillin-resistant Staphylococcus aureus using novel lytic phage-based magnetoelastic biosensors. Sens Actuators B, 210, 129-136
HiremathN, GuntupalliR, VodyanoyV, ChinBA, ParkMK. 2015; Detection of methicillin-resistant
Staphylococcus aureus using novel lytic phage-based magnetoelastic biosensors. Sens Actuators B. 210:129-136


Byeon HM, Vodyanoy VJ, Oh JH, Kwon JH, Park MK (2015) Lytic phage-based magnetoelastic biosensors for on-site detection of methicillin-resistant Staphylococcus aureus on spinach leaves. J Electrochem Soc, 162, B230-B235
ByeonHM, VodyanoyVJ, OhJH, KwonJH, ParkMK. 2015; Lytic phage-based magnetoelastic biosensors for on-site detection of methicillin-resistant
Staphylococcus aureus on spinach leaves. J Electrochem Soc. 162:B230-B235


Hiremath N, Chin BA, Park MK (2017) Effect of competing foodborne pathogens on the selectivity and binding kinetics of a lytic phage for methicillin-resistant Staphylococcus aureus detection. J Electrochem Soc, 164, B142-B146
HiremathN, ChinBA, ParkMK. 2017; Effect of competing foodborne pathogens on the selectivity and binding kinetics of a lytic phage for methicillin-resistant
Staphylococcus aureus detection. J Electrochem Soc. 164:B142-B146


Choi IY, Park JH, Gwak KM, Kim KP, Oh JH, Park MK (2018) Studies on lytic, tailed Bacillus cereus-specific bacteriophage for use in a ferromagnetoelastic biosensor as a novel recognition element. J Microbiol Biotechnol, 28, 87-94
ChoiIY, ParkJH, GwakKM, KimKP, OhJH, ParkMK. 2018; Studies on lytic, tailed
Bacillus cereus-specific bacteriophage for use in a ferromagnetoelastic biosensor as a novel recognition element. J Microbiol Biotechnol. 28:87-94.

SinghA, ArutyunovD, SzymanskiCM, EvoyS. 2012; Bacteriophage based probes for pathogen detection. Analyst. 137:3405-3421


Verma V, Harjai K, Chhibber S (2009) Characterization of a T7-like lytic bacteriophage of Klebsiella pneumoniae B5055: A potential therapeutic agent. Curr Microbiol, 59, 274-281
VermaV, HarjaiK, ChhibberS. 2009; Characterization of a T7-like lytic bacteriophage of
Klebsiella pneumoniae B5055: A potential therapeutic agent. Curr Microbiol. 59:274-281


Choi IY, Lee JH, Kim HJ, Park MK (2017) Isolation and characterization of a novel broad-host-range bacteriophage infecting Salmonella enterica subsp. enterica for biocontrol and rapid detection. J Microbiol Biotechnol, 27, 2151-2155
ChoiIY, LeeJH, KimHJ, ParkMK. 2017; Isolation and characterization of a novel broad-host-range bacteriophage infecting
Salmonella enterica subsp.
enterica for biocontrol and rapid detection. J Microbiol Biotechnol. 27:2151-2155


Oh JH, Park MK (2017) Recent trends in Salmonella Outbreaks and emerging technology for biocontrol of Salmonella using phages in foods: a review. J Microbiol Biotechnol, 27, 2075-2088
OhJH, ParkMK. 2017; Recent trends in
Salmonella Outbreaks and emerging technology for biocontrol of
Salmonella using phages in foods: a review. J Microbiol Biotechnol. 27:2075-2088


Yang H, Liang L, Lin S, Jia S (2010) Isolation and characterization of a virulent bacteriophage AB1 of Acinetobacter baumannii. BMC Microbiol, 10, 131-140
YangH, LiangL, LinS, JiaS. 2010; Isolation and characterization of a virulent bacteriophage AB1 of
Acinetobacter baumannii. BMC Microbiol. 10:131-140


AckermannHW, DuBowMS, GershmanM, Karska-WysockiB, KasatiyaSS, LoessnerMJ, Mamet-BratleyMD, RegueM. 1997; Taxonomic changes in tailed phages of enterobacteria. Arch Virol. 142:1381-1390


Jun JW, Giri SS, Kim HJ, Yun SK, Chi C, Chai JY, Lee BC, Park SC (2016) Bacteriophage application to control the contaminated water with Shigella. Sci Rep, 6, 22636
JunJW, GiriSS, KimHJ, YunSK, ChiC, ChaiJY, LeeBC, ParkSC. 2016; Bacteriophage application to control the contaminated water with
Shigella. Sci Rep. 6:22636


Faruque SM, Chowdhury N, Khan R, Hasan MR, Nahar J, Islam MJ, Yamasaki S, Ghosh AN, Nair GB, Sack DA (2003) Shigella dysenteriae type 1-specific bacteriophage from environmental waters in Bangladesh. Appl Environ Microbiol, 69, 7028-7031
FaruqueSM, ChowdhuryN, KhanR, HasanMR, NaharJ, IslamMJ, YamasakiS, GhoshAN, NairGB, SackDA. 2003;
Shigella dysenteriae type 1-specific bacteriophage from environmental waters in Bangladesh. Appl Environ Microbiol. 69:7028-7031


Jun JW, Kim JH, Shin SP, Han JE, Chai JY, Park SC (2013) Characterization and complete genome sequence of the Shigella bacteriophage pSf-1. Res Microbiol, 164, 979-986
JunJW, KimJH, ShinSP, HanJE, ChaiJY, ParkSC. 2013; Characterization and complete genome sequence of the
Shigella bacteriophage pSf-1. Res Microbiol. 164:979-986


Strauch E, Lurz R, Beutin L (2001) Characterization of a shiga toxin-encoding temperate bacteriophage of Shigella sonnei. Infect Immun, 69, 7588-7595
StrauchE, LurzR, BeutinL. 2001; Characterization of a shiga toxin-encoding temperate bacteriophage of
Shigella sonnei. Infect Immun. 69:7588-7595


Kim KH, Chang HW, Nam YD, Roh SW, Bae JW (2010) Phenotypic characterization and genomic analysis of the Shigella sonnei bacteriophage SP18. J Microbiol, 48, 213-222
KimKH, ChangHW, NamYD, RohSW, BaeJW. 2010; Phenotypic characterization and genomic analysis of the
Shigella sonnei bacteriophage SP18. J Microbiol. 48:213-222


Lee HY, Ku HJ, Lee DH, Kim YT, Shin HD, Ryu SY, Lee JH (2016) Characterization and genomic study of the novel bacteriophage HY01 infecting both Esherichia coli O157:H7 and Shigella flexneri potential as a biocontrol agent in food. PLoS One, 11, e0168985
LeeHY, KuHJ, LeeDH, KimYT, ShinHD, RyuSY, LeeJH. 2016; Characterization and genomic study of the novel bacteriophage HY01 infecting both
Esherichia coli O157:H7 and
Shigella flexneri potential as a biocontrol agent in food. PLoS One. 11 e0168985